凌晨!中国科大“霸屏”!
本文转自:合肥晚报
真·霸气如虎
开年仅10天
中国科大4项成果
在顶级科学杂志接连“霸屏”
北京时间2月10日凌晨 , 国际知名科学杂志《自然》发表了中国科学技术大学的3项重要科研成果 。采访人员获悉 , 开年仅10天 , 中国科大已有4项成果接连登上顶级科学杂志、期刊 , 给虎年开了个好头!
科学家建立蛋白质从头设计新方法

文章图片
文章图片
此次《自然》发表的3项成果中 , 包括该校刘海燕教授、陈泉副教授团队基于数据驱动原理 , 开辟出了一条全新的蛋白质从头设计路线 。该研究在蛋白质设计这一前沿科技领域实现了关键核心技术的原始创新 , 为工业酶、生物材料、生物医药蛋白等功能蛋白的设计奠定了坚实的基础 。
蛋白质是生命的基础 , 是生命功能的主要执行者 , 其结构与功能由氨基酸序列所决定 。目前 , 能够形成稳定三维结构的蛋白质 , 几乎全部是天然蛋白质 , 其氨基酸序列是长期自然进化形成 。在天然蛋白结构功能不能满足工业或医疗应用需求时 , 想要得到特定的功能蛋白 , 就需要对其结构进行设计 。近年来 , 国际上蛋白质从头设计的代表性工作主要使用天然结构片段作为构建模块来拼接产生人工结构 。然而 , 这种方法存在设计结果单一、对主链结构细节过于敏感等不足 , 显著限制了设计主链结构的多样性和可变性 。
中国科学技术大学相关团队长期深耕计算结构生物学方向的基础研究和应用基础研究 。施蕴渝院士是国内这一领域的开拓者 。刘海燕教授、陈泉副教授团队十余年来致力于发展数据驱动的蛋白质设计方法 。
该团队首先建立了给定主链结构设计氨基酸序列的ABACUS模型 , 进而发展了能在氨基酸序列待定时从头设计全新主链结构的SCUBA模型 。理论计算和实验证明 , 用SCUBA设计主链结构 , 能够突破只能用天然片段来拼接产生新主链结构的限制 , 从而显著扩展从头设计蛋白的结构多样性 , 甚至设计出不同于已知天然蛋白的新颖结构 。“SCUBA模型+ABACUS模型”构成了能够从头设计具有全新结构和序列的人工蛋白完整工具链 , 是RosettaDesign之外目前唯一经充分实验验证的蛋白质从头设计方法 , 并与之互为补充 。在论文中 , 团队报道了9种从头设计的蛋白质分子的高分辨晶体结构 , 其中5种蛋白质具有不同于已知天然蛋白的新颖结构 。
《自然》审稿人认为 , 这项工作中提出的方法具有足够的新颖性和实用性;从头设计蛋白质具有挑战性 , 本工作中6种不同蛋白质的高分辨率设计是一项重要成就 , 证明这种方法运行良好 。
四项成果接连登上
顶级科学杂志、期刊
2月10日 , 《自然》还同时发表了中国科大另2项重大成果 。
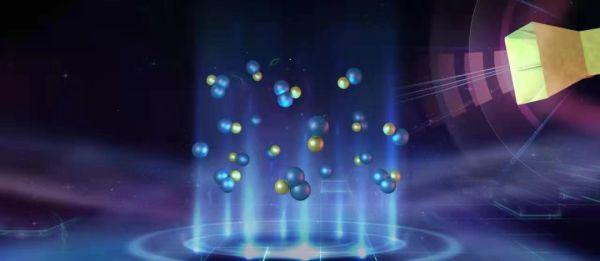
该校潘建伟、赵博等与中国科学院化学所白春礼小组合作 , 在超冷原子分子混合气中首次合成三原子分子 , 向基于超冷原子分子的量子模拟和超冷量子化学的研究迈出重要一步 。
文章图片
文章图片
从超冷原子和双原子分子混合气中利用射频场合成三原子分子的示意图 。
此外 , 该校陈仙辉、吴涛和王震宇等组成的团队 , 近日在笼目超导体CsV3Sb5中发现一种新型电子向列相 。该发现不仅为理解笼目结构超导体中电荷密度波与超导电性之间的反常竞争提供了重要实验证据 , 也为进一步研究关联电子体系中与非常规超导电性密切相关的交织序提供了新的研究方向 。
- “观·味”中国年|快递业春节包裹量增16%达7.5亿, 掀“不打烊”活动应对“小高潮”
- DMV最新自动驾驶报告出炉,中国玩家名列前茅
- 化学物理重大突破!中国首次在超冷原子分子混合气中实现三原子分子的量子相干合成
- “在中国同行面前,我们自愧不如!”
- 嫦娥五号传回好消息!中国公开一项神秘信息,全新发现解决难题
- 《中国航天科技活动蓝皮书(2021》发布,中国空间站年底前将全面建成
- 冬奥会史上首次实现火炬零碳排放!背后是中国航天力量护航
- 中国首次深水犁式挖沟机海试成功
- 长沙市民买“無印良品”,收款方却是数码公司!商家:我们是中国的无印良品
- 3D打印建造技术日趋成熟
