世界首张小鼠“扰动图谱”揭秘基因功能!上海科技大学池天团队成果登上《细胞》
本文转自:文汇报

文章图片
文章图片
▲上科大生命科学与技术学院池天团队 。(前排左1 陈玉鑫 前排左3 毛少帅 二排左5 荆征宇 二排左6 池天 二排左7 张校铭 三排 刘波)
昨天(7月22日) , 上海科技大学生命学院池天课题组在《细胞》(Cell)杂志在线发表论文 , 报道了一种崭新的小鼠基因打靶技术iMAP , 快速鉴定了90个基因在39种组织的基本功能 , 构建了世界首张小鼠微型“扰动图谱” 。
早在2001年 , 国际人类基因组计划就公布了人类基因组序列 。但目前为止 , 约两万个哺乳动物蛋白编码基因在500多种细胞中的功能仍不为人知 , 这严重妨碍了疾病的诊断和治疗 。
要系统性地解码全部基因在全部细胞中的功能 , 必须将其分别在各种细胞中敲除(扰动)再鉴定其细胞表型 , 即描绘完整的“扰动图谱” 。
“扰动图谱”的完成 , 将成为生物医学研究的分水岭:此后 , 要探索任何基因的基本功能 , 将会像查找基因序列一样简单 。但用传统的基因打靶方法 , 无法快速有效地描绘扰动图谱 , 成为功能基因组学领域久攻不下的瓶颈 。
池天课题组里程碑式的成果为描绘完整扰动图谱迈出了从0到1的第一步 。
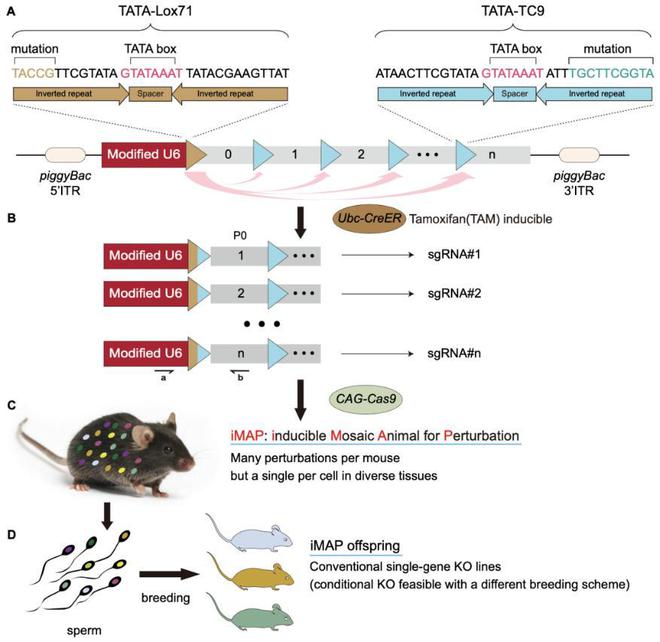
iMAP融合了Cre-loxP和CRISPR-Cas9技术 , 其核心组成部分是一个转基因序列 , 它由U6启动子和下游一串gRNA表达单位构成 。转基因通常只表达第?个sgRNA , 而利用药物(他莫昔芬)激活Cre , 导致转基因重组 , 可使其余的sgRNA也得以表达 , 但每个细胞只随机表达其中?个 。
这些sgRNA可在Cas9的帮助下敲除相应的靶基因 , 从而将小鼠转化为嵌合体 。这种嵌合体可立即用于描绘扰动图谱 , 也可迅速、廉价地繁育出多种传统的单基因敲除品系 。该品系不但可用于特定靶基因的后续深入研究 , 更可用于个体水平的表型筛选 , 从而与嵌合体优势互补 , 共同加速基因解码 。
文章图片
文章图片
▲iMAP原理
iMAP诞生的背后是一场长达八年、历经六届学生前赴后继努力的科学“马拉松” 。2014年 , 上海科技大学生命学院教授池天还在耶鲁大学 , 当时CRISPR基因编辑技术已问世 , 而池天又熟悉Cre-loxP 。
他突发奇想 , 试图融合这两种技术 , 但同事并不看好 。“有人不屑一顾 , 有人直呼疯狂 , 大多心存疑虑 。”池天回忆 , “这也不奇怪 , 因为iMAP太新了 , 是0到1 , 没有主流技术可以直接对标 。就如同当年PCR概念萌芽后 , 甚至初步结果出来后 , 仍然在Cetus公司内部遭到冷遇 , 这好像不可思议 , 其实不难理解 。”
iMAP的研发过程充满了艰辛和曲折 。“我们踩了很多坑 , 有些坑很诡异 。”为使同行避免重蹈覆辙 , 这些失败的实验 , 部分已于2年前以预印本形式发表 。
经Ravinder Kaundal (耶鲁大学博士后)和陈玉鑫、毛少帅、刘波(分别是上科大生命学院2014、2015、2016级研究生)等的努力 , 到2018级博士生荆征宇加入课题组时 , 终于曙光初照 , 但道路依然坎坷 。
除了学生非常给力 , 学校的大力支持也让池天的研究如鱼得水 。
“我在耶鲁启动过其他数个像iMAP一样的高风险课题 , 但都因经费原因夭折了 。而上科大鼓励教授勇于坐冷板凳 , 诺奖得主James Rothman也表示上科大适合我 。所以我对学生说 , 课题必须做到底 , 论文非顶刊不发 。”池天说 。
- 迷你世界马红俊激活码分享(2022马红俊皮肤兑换码最新)
- 迷你世界奥斯卡兑换码是什么(2022最新奥斯卡皮肤激活码分享)
- 全世界有20多亿家庭在使用中国的家电产品
- 总台记者看世界丨从航空先驱“杜蒙特”到垂直起降“飞行汽车”,巴西人一直“想上天”
- 迷你世界2022年7月22日最新激活码(2022迷你世界7.22cdk兑换码)
- 上海市国家专精特新“小巨人”企业荣获世界知识产权组织(WIPO)全球奖优胜企业
- 迷你世界2022年7月21日最新激活码(2022迷你世界7.21cdk兑换码)
- 梦幻西游龙潭虎穴通关攻略(画中大冒险世界四100%探索度)
- 我国数字经济规模超45万亿元!数年稳居世界第二
- 国际知名数学家丘成桐贵州行|让贵州成为世界数据智库中心
