AI角逐生命科学的新风口 中国暂时占住了( 二 )
而且解蛋白质结构之题 , 远远难于基因 。“蛋白质结构测定与基因组测序最大区别在于 , 基于组序列是一维的、线性的 , 信息仅有4种可能的代码(A、C、T、G);而蛋白质结构是三维的 , 每个原子的坐标在XYZ三个方向上都可以是任意的数值 。因此 , 基因组测序无论测什么物种的基因组 , 本质上都是在做同一件事 , 可以通过机器进行自动化、规模化的操作 , 而蛋白质结构测定则是不同的课题 , 科学家始终没有找到批量处理的方法 。”叶盛说 。
在过去几十年的研究中 , 结构生物学家为探测蛋白质结构发展出三大实验手段:X射线晶体学、核磁共振和冷冻电镜 。但实验方法成本高、周期长 。目前人类已知有数十亿蛋白质序列 , 还原出结构的却只有十几万 。生物学发展因此颇受掣肘 。
人工智能的发展为解决这一问题带来了一些新的可能 。在2020年的国际蛋白质结构预测顶级竞赛 CASP14上 , Google DeepMind推出AlphaFold2(简称AF2)预测的大部分单体蛋白质结构准确度与实验足够接近 , 远超此前所有方法 。这一结果也标志着蛋白质单体结构的问题在一定意义上得到了解决 。中国结构生物学家施一公对此的评价为“这是人工智能对科学领域的最大一次贡献 。”结构生物学家Petr Leiman表示:“我用价值一千万美元的电镜努力了好几年 , AF2竟然一下就算出来了 。”
除了AlphaFold2 , 另外一个被高度关注的AI算法是华盛顿大学医学院蛋白质设计研究所的贝克团队 , 有人说因为DeepMind的成功触动贝克团队重视AI , 于是其联合很多大学与机构研发了一款基于深度学习的蛋白质预测新工具 RoseTTAFold , 并在预测蛋白质结构上取得了媲美AF2的超高准确率 , 而且速度更快、所需要的计算机处理能力也较低 。
【AI角逐生命科学的新风口 中国暂时占住了】总之 , AlphaFold2与RoseTTAFold是AI预测蛋白质结构的两个“明星” , 破解了出现五十年之久的蛋白质分子折叠问题 , 打开了人类预测蛋白质结构的另一扇窗 。今年7月 , DeepMind团队与贝克团队分别开源了他们各自得算法 , 这给全球研究机构进行蛋白质结构预测带来福音 , 大大降低研究蛋白质结构的门槛 , 将帮助科研人员弄清引发某些疾病的机制 , 并为设计药物、农作物增产 , 以及可降解塑料的“超级酶”等的研发铺平道路 。
蛋白质结构预测的中国智慧
AlphaFold2与RoseTTAFold首次展现了AI在蛋白质结构预测领域的巨大想象力 , 意识到它可能带来划时代的意义 , 中国的AI公司、研究团队同样加入了这场解码生命难题之战 。
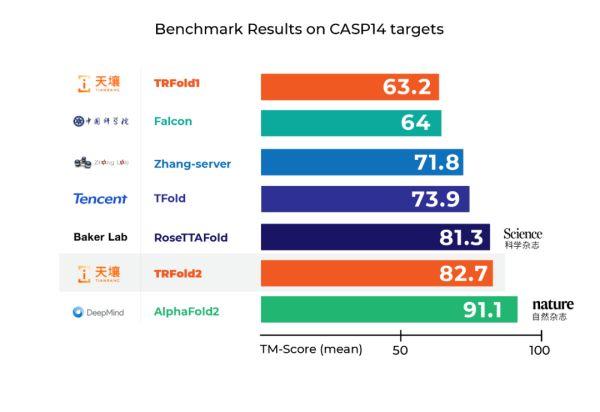
文章图片
文章图片
或许有人会问 , AF2与 RoseTTAFold都已经开源 , 中国有必要再自主研发蛋白质结构预测的算法吗?答案是当然必须 , 开源带来的也并不全是便利 , 占据自主研发的高地才能避免在未来的科技竞争中卡脖子 。虽然 DeepMind开源了 AF2 模型的推理代码 , 但其训练代码并未公布 , 且模型不可商用 。
无法深入过程 , 就无法对不同的方法优势进行量化 , 也不能将最有价值的创新进行更大范围的应用 。天壤创始人薛贵荣认为:“AF2的成功是蛋白质结构预测方向的重大突破 , 但是围绕蛋白质结构功能问题且能够达到实际落地应用准确度要求的AI算法的开发才刚刚开始 , 没有训练模型经验 , 或者没有具备能够训练出AF2结果的能力是无法把该技术推进解决更深层次问题的 。”
在这一点上 , 中科院院士、北京大学教授、深势科技首席科学顾问鄂维南表达了与薛贵荣一致的观点:“尽管DeepMind开源了推理代码 , 但模型的训练技术才是核心竞争力 。”
- 人类与AI如何共处?诺奖科学家、将棋天才、“低欲望社会”提出者的不同解答
- 冬奥在即!“AI教练”、滑雪机器人,沪上高校科技助力“冰雪精灵”!
- 智能科技傍身,讯飞智能录音笔SR101助力学习生活
- “集五福”玩出新高度,云宠宝的这波黑科技营销,直接天花板了!
- 冬奥防疫“黑科技”上线,45分钟完成空气气溶胶病毒检测
- 微软、动视暴雪并购电话会实录:元宇宙、云游戏,开启新一轮全球硬科技战事
- 细节里都是绿色科技
- 电动牙刷哪个牌子好?实用科普不同价位的电动牙刷如何选?
- 「科普」世界上有哪些类型的海洋钻井平台?
- 我国首次实现低轨宽带卫星批产;研究称奥密克戎在陶瓷表面存活时间最短丨科技早新闻
